自己組織化マップ (Self-Organizing Map; SOM)#

# 必要なパッケージをimport
from tqdm.auto import tqdm,trange
import numpy as np
import matplotlib.pyplot as plt
import matplotlib_fontja
matplotlibで日本語を使うための準備
# Python 3.12以降
try:
import matplotlib-fontja
except:
!pip install matplotlib-fontja
import matplotlib_fontja
# Python 3.11以前
try:
import japanize_matplotlib
except:
!pip install japanize_matplotlib
import japanize_matplotlib
近傍関数を簡単のためにシンプルにしたバージョン#
実験設定#
ハイパーパラメータ を定義。
# 潜在空間の範囲を指定(変数名はもっといい名付け方があると思います...)
n_xaxis = 30
n_yaxis = 30
# 潜在空間上のグリッドにはそれぞれ、ニューロンが配置されます。このニューロンが持っている参照ベクトルの次元数を定義。入力データの特徴数と同じ値です。
n_channels = 3
# 最大学習回数
max_iter = 100
# 学習率。αやlr(leraning_rate)と言われることが多いです。
alpha = 0.08
学習可能パラメータ を準備。
# 二次元の配列のそれぞれの要素がベクトルであるイメージ。つまり実際には三次元配列になる。
weight = np.random.random([n_xaxis,n_yaxis,n_channels])
# 定義した学習可能パラメータをheatmapとして可視化してみよう。
plt.title("SOMの学習可能パラメータ(初期状態)")
plt.imshow(weight)
plt.show()
学習に用いるサンプルデータを準備#
デモンストレーションを行うために、訓練に用いるサンプルデータを作ります。ここでは、RGBの特徴を持ったベクトルを100個作りましょう。
demo_data = np.random.random([100, 3])
plt.figure(figsize=(40,400))
plt.title("SOMに学習させるサンプルデータ")
plt.imshow(demo_data.T)
plt.show()

訓練stepの作成#
訓練データを一つ一つ受け取り、パラメータを更新する関数を作成する。
def som(color_vec, weight):
"""Self-Organizing Mapの学習可能パラメータ(weight)の更新を行う関数。
データを一つ一つ受け取り、最も類似度の高いニューロンとその周辺(前後左右各2マス分)のパラメータを更新する。
ただし、簡単のために近傍関数はステップ関数にしている。
"""
# 入力データ(color_vec)と最も近い座標を特定する。
min_index = np.argmin(((weight - color_vec)**2).sum(axis=2))
# ただし、二次元座標が欲しいので変換する。
_, n_yaxis, _ = weight.shape
mini = int(min_index / n_yaxis)
minj = int(min_index % n_yaxis)
# 選ばれたニューロンの近傍(前後左右2マス)の重みを更新する。
for i in range(-2,3): # -2, -1, 0, 1, 2
for j in range(-2,3): # -2, -1, 0, 1, 2
try:
weight[mini+i,minj+j] += alpha * (color_vec - weight[mini+i,minj+j])
except:
pass
return weight
訓練ループの作成と実行#
訓練が長時間になる可能性があるので、ここではtrangeを使う。これはプログレスバーの表示とrange関数の機能を両方持った関数。
for time in trange(max_iter):
for color_vec in demo_data:
weight = som(color_vec, weight)
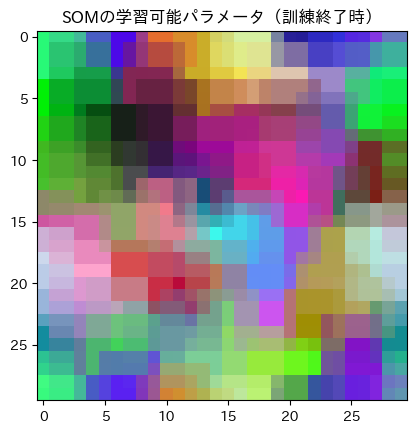
plt.title("SOMの学習可能パラメータ(訓練終了時)")
plt.imshow(weight,interpolation='none')
plt.show()
課題(1)#
デモデータをSOMで訓練して作成した潜在空間を画像として保存し、moodleの(課題3)に提出せよ。
課題(2)#
近傍関数を以下の要請に従うように作成せよ。
argumentsとして「入力データと最も近いニューロンのindex」と「潜在空間のshape」を受け取る
そのニューロンと全てのニューロンとの距離を計算する
距離行列を0~1に収まるように正規化する
類似度行列=1- 距離行列
類似度行列をreturnする
これをもとに上記のsomプログラムを修正せよ。(パラメータ更新の際に、全てのニューロンに対する近傍関数の値と更新値との要素積を取ればいいはず)
最後に、作成した潜在空間を画像として表示せよ。
ヒント#
ヒント1 :
以下のように実装できるはず。(get_axis_matrixと正規化関数を作成する必要がある)
def neighborhood(best_index, latent_shape):
潜在空間での座標 = get_axis_matrix(*latent_shape) # 例として(5,5)
距離 = np.sum((潜在空間での座標 - best_index)**2, axis=2) # 引き算して
正規化した距離 = 正規化(距離)
類似度 = 1- 正規化した距離
return 類似度
ヒント2:
正規化は
(距離-距離.min()) / (距離.max() - 距離.min())
で実装できる
ヒント3:
get_axis_matrixを以下に示す。
def get_axis_matrix(x_length, y_length):
"""
潜在空間のshapeを渡すと、各ニューロンの座標を入れた三次元配列を返す
"""
axis_matrix = []
for i in range(x_length):
axis_matrix.append([])
for j in range(y_length):
axis_matrix[-1].append([i,j])
return np.array(axis_matrix)
ヒント4:
近傍関数を以下に示す。
def neighborhood(best_index, latent_shape):
潜在空間での座標 = get_axis_matrix(*latent_shape) # 例として5,5
距離 = np.sum((潜在空間での座標 - best_index)**2, axis=2) # 引き算して
正規化した距離 = (距離-距離.min()) / (距離.max() - 距離.min())
類似度 = 1- 正規化した距離
return 類似度
print(neighborhood([1,2], [5,5]).shape)
neighborhood([1,2], [5,5])
(5, 5)
array([[0.61538462, 0.84615385, 0.92307692, 0.84615385, 0.61538462],
[0.69230769, 0.92307692, 1. , 0.92307692, 0.69230769],
[0.61538462, 0.84615385, 0.92307692, 0.84615385, 0.61538462],
[0.38461538, 0.61538462, 0.69230769, 0.61538462, 0.38461538],
[0. , 0.23076923, 0.30769231, 0.23076923, 0. ]])

